El equipo del profesor Dominik Niopek del Instituto de Farmacia y Biotecnología Molecular (IPMB) de la Universidad de Heidelberg ha desarrollado un nuevo modelo de inteligencia artificial llamado ProDomino, que predice la forma de ensamblaje de subunidades proteicas para diseñar proteínas artificiales con funciones específicas. Los resultados de esta investigación se han publicado en la revista Nature Methods, proporcionando nuevas herramientas de investigación para los campos de la biotecnología y la medicina.
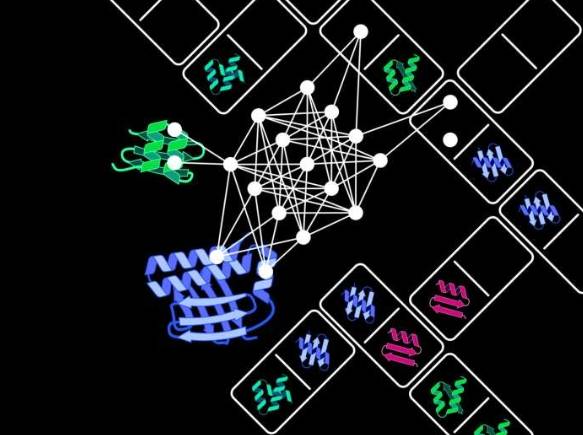
Las proteínas son las "máquinas moleculares" de las células, compuestas por múltiples dominios (subunidades) responsables de funciones clave como el metabolismo energético y la síntesis de material genético. Inspirados en la evolución de proteínas en la naturaleza, el equipo de Niopek utilizó un modelo de IA para simular la reorganización de dominios, desarrollando la herramienta de código abierto ProDomino. Esta herramienta analiza más de 100.000 conjuntos de datos de proteínas para predecir las combinaciones óptimas de dominios y construir nuevas proteínas con funciones controlables.
El equipo ya ha aplicado ProDomino en casos prácticos, como la combinación de dominios sensibles a químicos con el sistema CRISPR-Cas para generar herramientas de edición génica regulables. El líder del proyecto, el Dr. Jan Mathony, indicó: "Estas proteínas híbridas pueden activarse o desactivarse con precisión, mejorando la seguridad de la edición genómica". Actualmente, el modelo ha sido validado en múltiples proteínas y está disponible para investigadores globales.
El profesor Niopek señaló: "ProDomino no solo simplifica la preparación de proteínas artificiales, sino que también mejora la precisión del diseño, abriendo nuevos caminos para aplicaciones biomédicas". En el futuro, esta tecnología podría impulsar el desarrollo de métodos de tratamiento personalizados.